Nelson H. Morgon1 e Aguinaldo R. de Souza2
1Instituto de Química e Centro de Computação em Engenharia e Ciências – Universidade Estadual de Campinas, Campinas, SP.
2Faculdade de Ciências da Universidade Estadual Paulista, Bauru, SP.
Quando estamos em estado febril devido a alguma enfermidade ,como uma gripe ou um simples resfriado, fazemos uso de algum medicamento oriundo de um fármaco encontrado na natureza ou produzido em laboratório. Ao ingerirmos esse medicamento o objetivo é recuperarmos a nossa saúde, no entanto alguns fenômenos muito interessantes ocorrem no nosso organismo em nível molecular que atraem o interesse dos pesquisadores. A atenção está voltada, principalmente no estudo das interações entre os medicamentos que ingerimos e as biomoléculas do nosso corpo como as proteínas e o DNA.

Cristais da proteína HSA.
Fonte: (https://www.imperial.ac.uk/people/s.curry)
Dentre um número muito grande de proteínas encontradas no nosso corpo, uma delas é essencial no transporte, ou na farmacocinética, de remédios e de outras moléculas de interesse biológico: a Albumina do Soro Humano, ou HSA (do inglês Human Serum Albumin). Esta proteína, produzida no fígado, é a mais abundante no sangue, e exerce muitas funções vitais no nosso organismo, como a regulação da acidez (pH) do nosso sangue. Outra função importante dessa proteína é o transporte dos hormônios da glândula tireoide, conhecidos como T3 e T4. A HSA é uma proteína globular, na forma de coração, que apresenta a propriedade de ligar-se a várias moléculas tanto de origem do nosso próprio corpo (endógenas) ou externas a ele (exógenas). O estudo da ligação de moléculas à proteína HSA é realizado através da obtenção de pequenos cristais contendo tanto a proteína como o fármaco.
A partir destes cristais e da técnica de difração de raios-X podemos obter informações valiosas sobre a orientação de um fármaco dentro da proteína. Esta informação é essencial para o desenvolvimento de novos fármacos para o tratamento de enfermidades e no alívio de sintomas advindos de doenças ou síndromes. No entanto, para alguns fármacos, após a interação com a proteína HSA, não é possível, e em alguns casos é impossível a obtenção desses cristais. Deste modo lança-se mão de outros recursos, como a simulação computacional dessas estruturas químicas. Assim, com o aparecimento, nas últimas décadas, de computadores cada vez mais velozes e com maior capacidade de armazenamento de informações, e também com o desenvolvimento de programas cada vez mais inteligentes, hoje em dia é possível o estudo de sistemas cada vez mais complexos. Dentre estes problemas está o estudo das interações entre fármacos e a proteína HSA. Estes estudos são conhecidos na comunidade científica como Modelagem Molecular. A característica principal desses estudos é o caráter preditivo das propriedades eletrônicas e moleculares dos sistemas envolvendo, por exemplo, estudo das propriedades isoladas dos fármacos e da proteína HSA, bem como de suas interações.
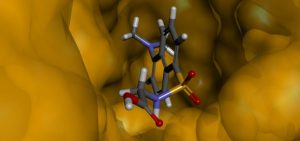
Vista da estrutura da cavidade do sítio de interação da HSA com o aminoácido dansilglicina.
Os cálculos matemáticos envolvidos nestes estudos são relativamente custosos do ponto de vista computacional, ou seja, demoram muito tempo, e por isso o nosso grupo de pesquisa tem conseguido estudar os problemas envolvendo a interação entre diferentes fármacos e a proteína HSA graças à infraestrutura disponível no Center for Computing in Engeneering & Sciences (CCES). Os resultados obtidos na Modelagem Molecular permitem a obtenção de informações que servem como subsídios na interpretação dos resultados obtidos experimentalmente (nos laboratórios), além da possibilidade de predição das propriedades químicas e físicas do sistema fármaco e proteína. Na figura acima, por exemplo, apresentamos um dos resultados obtidos nos cálculos computacionais ,que mostra a molécula de um fármaco no interior da proteína HSA.
Artigos científicos relacionados:
Venturini, D.; Pastrello, B.; Zeraik, M. L.; Pauli, I.; Andricopulo, A. D.; Silva-Filho, L. C.; Bolzani, V. S.; Morgon, N. H.; Souza, A. R.; Ximenes, V. F. Experimental, DFT and docking simulations of the binding of diapocynin to human serum albumin: induced circular dichroism. RSC Adv. 5(76), pp. 62220-62228, 2015.
De Souza, A. R.; Ximenes, V. F.; Morgon, N. H. Study of relative stability of tautomeric forms of phenylbutazone and calculation of UV-Vis and ECD spectra. Rev. Virtual. Quím. 8(2), pp. 525-535, 2016.
Venturini, D.; De Souza, A. R.; Caracelli, I.; Morgon, N. H.; SILVA-FILHO, L. C.; Ximenes, V. F. Induction of axial chirality in divanillin by interaction with bovine serum albumin. PLoS One, 12, pp. e0178597, 2017.
Ximenes, V. F.; Morgon, N. H., De Souza, A. R. Solvent-dependent inversion of circular dichroism signal in naproxen: An unusual effect!. Chirality 30, pp. 1049-1053, 2018.
De Souza, A. R.; Boza, I. A. F.; Ximenes, V. F.; Yoguim, M. I.; Dávila-Rodrigues, M. J. Morgon, N. H.; Caracelli, I. Elucidação da quiralidade induzida na molécula dansilglicina na complexação com a proteína albumina do soro humano (HSA). Quím. Nova, 42(2), pp. 135-142, 2019.
Sousa, I. L., Ximenes, V. F.; De Souza, A. R., Morgon, N. H. Solvent-Induced Stokes’ Shift in DCJTB: Experimental and Theoretical Results. J. Mol. Struct. (in press).

